利用SimBiology模型分析仪计算NCA参数并拟合模型到PK/PD数据gydF4y2Ba
这个例子展示了如何进行非区隔分析来计算NCA参数和估计肿瘤生长模型gydF4y2Ba[1]gydF4y2Ba参数从实验数据中采用非线性回归gydF4y2BaSimBiology模型分析gydF4y2Ba应用程序。gydF4y2Ba
肿瘤的生长模型gydF4y2Ba
本例中使用的模型是SimBiologygydF4y2Ba®gydF4y2BaSimeoni等人的药代动力学/药效学(PK/PD)模型的实施。量化了抗癌药物对肿瘤生长动力学的影响gydF4y2Ba在活的有机体内gydF4y2Ba动物研究。药物的药代动力学由双室模型描述与静脉给药和线性消除(gydF4y2Ba柯gydF4y2Ba)gydF4y2Ba中央gydF4y2Ba隔间。肿瘤生长是一个双阶段的过程,最初是指数增长,然后是线性增长。增殖肿瘤细胞的生长速率用gydF4y2Ba
lgydF4y2Ba0gydF4y2Ba,gydF4y2BalgydF4y2Ba1gydF4y2Ba, Ψ为肿瘤生长参数,gydF4y2BaxgydF4y2Ba1gydF4y2Ba是增殖的肿瘤细胞的重量,和gydF4y2BawgydF4y2Ba为肿瘤总重量。在没有任何药物的情况下,肿瘤只由增殖细胞组成,也就是说,gydF4y2BawgydF4y2Ba=gydF4y2BaxgydF4y2Ba1gydF4y2Ba.在抗癌剂的存在下,一小部分增殖细胞转化为非增殖细胞。这种转化的速率被认为是血浆中药物浓度和疗效因子的函数gydF4y2BakgydF4y2Ba2gydF4y2Ba.nonproliferating细胞gydF4y2Bax2gydF4y2Ba经历一系列的过渡阶段(gydF4y2Bax3gydF4y2Ba而且gydF4y2Bax4gydF4y2Ba),并最终从系统中清除。将过渡舱的流动建模为速率常数的一阶过程gydF4y2BakgydF4y2Ba1gydF4y2Ba.gydF4y2Ba
SimBiology模型对肿瘤生长的药效学进行了以下调整:gydF4y2Ba
而不是把肿瘤重量定义为gydF4y2Bax1gydF4y2Ba,gydF4y2Bax2gydF4y2Ba,gydF4y2Bax3gydF4y2Ba,gydF4y2Bax4gydF4y2Ba,模型通过命名的反应来定义肿瘤的重量gydF4y2Ba增加gydF4y2Ba,gydF4y2Ba
零→tumor_weightgydF4y2Ba,与反应速率gydF4y2Ba .gydF4y2Batumor_weightgydF4y2Ba为肿瘤总重量,gydF4y2BaxgydF4y2Ba1gydF4y2Ba是增殖的肿瘤细胞的重量,和gydF4y2BalgydF4y2Ba0gydF4y2Ba,gydF4y2BalgydF4y2Ba1gydF4y2Ba为肿瘤生长参数。gydF4y2Ba
同样,该模型通过所命名的反应来定义肿瘤重量的减少gydF4y2Ba衰变gydF4y2Ba,gydF4y2Ba
tumor_weight→零gydF4y2Ba,与反应速率gydF4y2BakgydF4y2Ba1gydF4y2Ba*gydF4y2BaxgydF4y2Ba4gydF4y2Ba.常数gydF4y2BakgydF4y2Ba1gydF4y2Ba是转发速率参数,和gydF4y2BaxgydF4y2Ba4gydF4y2Ba是肿瘤重量转运减少系列中的最后一种。gydF4y2Ba柯gydF4y2Ba是间隙和中央隔间体积的函数:gydF4y2Ba
克= Cl_Central /中央gydF4y2Ba.gydF4y2Ba

PK / PD数据描述gydF4y2Ba
实验(合成)数据包含了8名患者对三种反应的测量:测量中心室、外周室的药物浓度和测量肿瘤重量。数据还包含给药信息,每个患者在第7天接受静脉注射。gydF4y2Ba
该数据集包含以下列。gydF4y2Ba
IDgydF4y2Ba——病人idgydF4y2Ba
时间gydF4y2Ba-测量的时间gydF4y2Ba
CentralConcgydF4y2Ba-药物集中在中心隔间gydF4y2Ba
PeripheralConcgydF4y2Ba-外周室药物浓度gydF4y2Ba
剂量gydF4y2Ba-每个患者的给药信息gydF4y2Ba
k1gydF4y2Ba而且gydF4y2BaCl_CentralgydF4y2Ba-变量列,其中包含特定于组的参数值gydF4y2Bak1gydF4y2Ba而且gydF4y2BaCl_CentralgydF4y2Ba.gydF4y2Ba
南gydF4y2Ba在没有测量或没有给予剂量的情况下使用数值。gydF4y2Ba
负荷肿瘤生长模型及数据gydF4y2Ba
在命令行中输入:gydF4y2Ba
simBiologyModelAnalyzer (gydF4y2Ba“tumor_growth_fitPKPD.sbproj”gydF4y2Ba);gydF4y2BaSimBiology模型分析gydF4y2Ba打开。在gydF4y2Ba浏览器gydF4y2Ba窗格中,gydF4y2Ba模型gydF4y2Ba文件夹中包含gydF4y2Ba
肿瘤的生长模型gydF4y2Ba和gydF4y2BaData1gydF4y2Ba文件夹包含实验数据和剂量信息。gydF4y2Ba对数据列进行分类,以便此类变量分类可用于gydF4y2Ba适合gydF4y2Ba在后面的示例中进行编程。该应用程序根据需要执行自动分类(如gydF4y2BaIDgydF4y2Ba,gydF4y2Ba时间gydF4y2Ba,gydF4y2Ba剂量gydF4y2Ba列)。但对于测量的响应数据列如gydF4y2BaCentralConcgydF4y2Ba,需要手动将它们分类为因变量。为此,首先打开如下所示的数据表。在gydF4y2Ba浏览器gydF4y2Ba窗格中,展开gydF4y2BaData1gydF4y2Ba文件夹,双击gydF4y2BaDatasheet1gydF4y2Ba.gydF4y2Ba
在gydF4y2BaData1gydF4y2Ba表,双击gydF4y2Ba分类gydF4y2Ba下gydF4y2BaCentralConcgydF4y2Ba.选择gydF4y2Ba
依赖gydF4y2Ba.重复相同的过程gydF4y2BaPeripheralConcgydF4y2Ba而且gydF4y2BaTumorWeightgydF4y2Ba.你可以离开gydF4y2Bak1gydF4y2Ba而且gydF4y2BaCl_CentralgydF4y2Ba列设置。gydF4y2Ba请注意gydF4y2Ba
该应用程序已经自动分类:gydF4y2Ba
的gydF4y2BaIDgydF4y2Ba列gydF4y2Ba集团gydF4y2Ba(分组变量)。gydF4y2Ba
的gydF4y2Ba时间gydF4y2Ba列gydF4y2Ba独立的gydF4y2Ba(独立变量)。gydF4y2Ba
的gydF4y2Ba剂量gydF4y2Ba列gydF4y2Badose1gydF4y2Ba(定量变量)。如果有一个以上的剂量列,它们可以分类为gydF4y2Badose2gydF4y2Ba,gydF4y2Badose3gydF4y2Ba,等等。gydF4y2Ba

可视化实验数据gydF4y2Ba
加载数据之后,就可以可视化测量的响应了。gydF4y2Ba
在gydF4y2Ba浏览器gydF4y2Ba窗格中,单击gydF4y2BaData1gydF4y2Ba.gydF4y2Ba
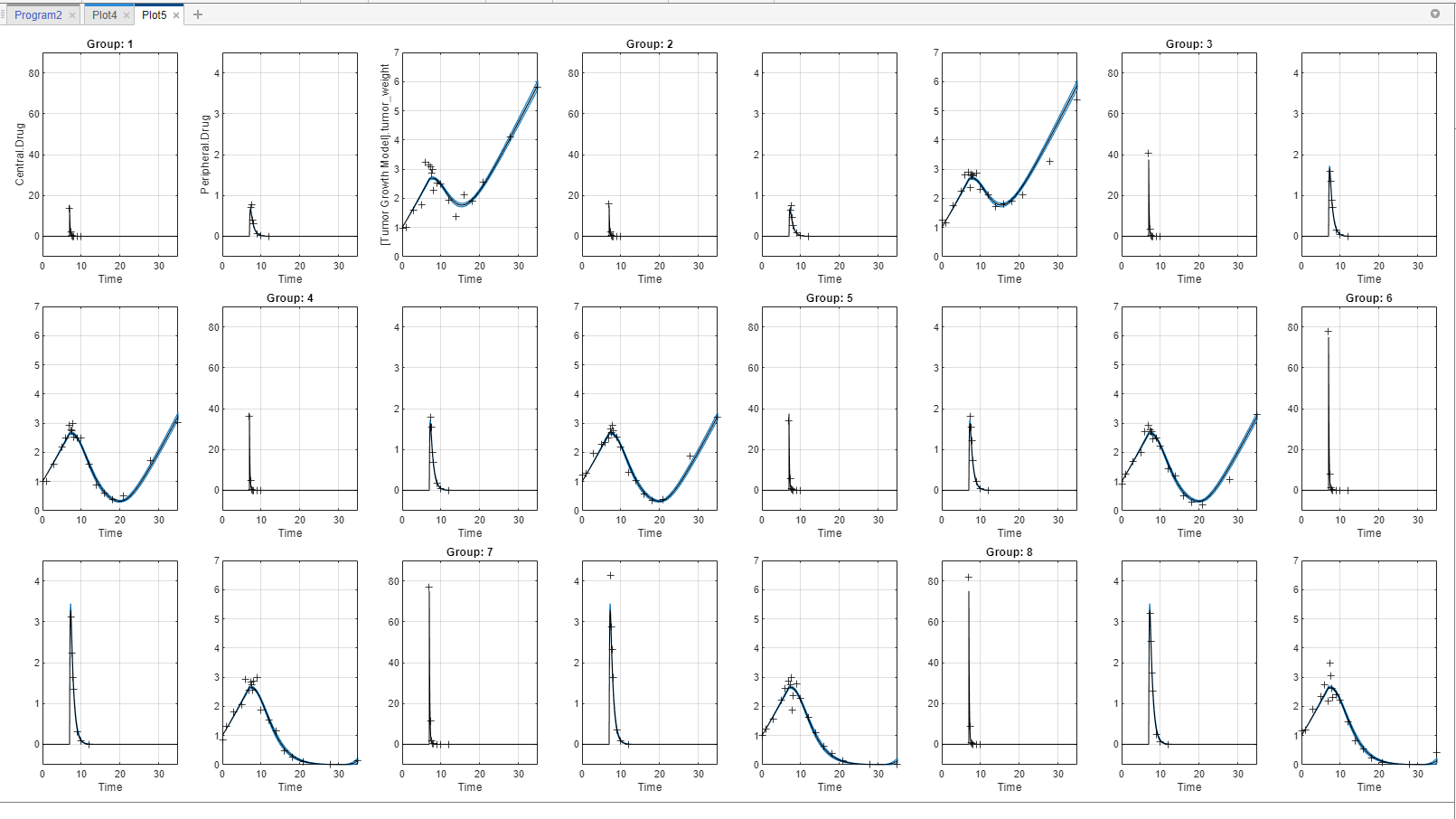
在gydF4y2Ba首页gydF4y2Ba选项卡,gydF4y2Ba情节gydF4y2Ba部分,单击gydF4y2Ba时间gydF4y2Ba情节。该应用程序生成了所有三种响应的时间图,即:gydF4y2BaCentralConcgydF4y2Ba,gydF4y2BaPeripheralConcgydF4y2Ba,gydF4y2BaTumorWeightgydF4y2Ba.gydF4y2Ba

在默认时间图中,gydF4y2Ba反应gydF4y2Ba与测量的响应相对应,并使用不同的线样式绘制。gydF4y2Ba场景gydF4y2Ba指数据中的不同组(8例患者),用不同颜色绘制。gydF4y2Ba
提示gydF4y2Ba
绘图由当前存在于应用程序工作区中的数据支持。图不是快照。当数据(实验数据或模拟结果)被删除或更改时,图也会根据基础数据的更改进行更新。gydF4y2Ba
定制数据可视化gydF4y2Ba
本节中的步骤是可选的,对于拟合不是必需的。你可以自定义情节,让它更清晰。例如,您可以绘制PD数据(gydF4y2BaTumorWeightgydF4y2Ba)与PK数据(gydF4y2BaCentralConcgydF4y2Ba而且gydF4y2BaPeripheralConcgydF4y2Ba).为此,创建两个不同的组(gydF4y2Ba集gydF4y2Ba),其中第一组只包含gydF4y2BaTumorWeightgydF4y2Ba第二组包含gydF4y2BaCentralConcgydF4y2Ba而且gydF4y2BaPeripheralConcgydF4y2Ba.gydF4y2Ba
右键单击gydF4y2BaTumorWeight(克)gydF4y2Ba在gydF4y2Ba反应gydF4y2Ba表并选择gydF4y2Ba
创建新组gydF4y2Ba.这个应用程序创建gydF4y2Ba组1gydF4y2Ba而且gydF4y2Ba组2gydF4y2Ba.gydF4y2Ba组1gydF4y2Ba只包含gydF4y2BaTumorWeightgydF4y2Ba,现在它被绘制在不同的轴上gydF4y2Ba组2gydF4y2Ba,其中包含gydF4y2BaCentralConcgydF4y2Ba而且gydF4y2BaPeripheralConcgydF4y2Ba.gydF4y2Ba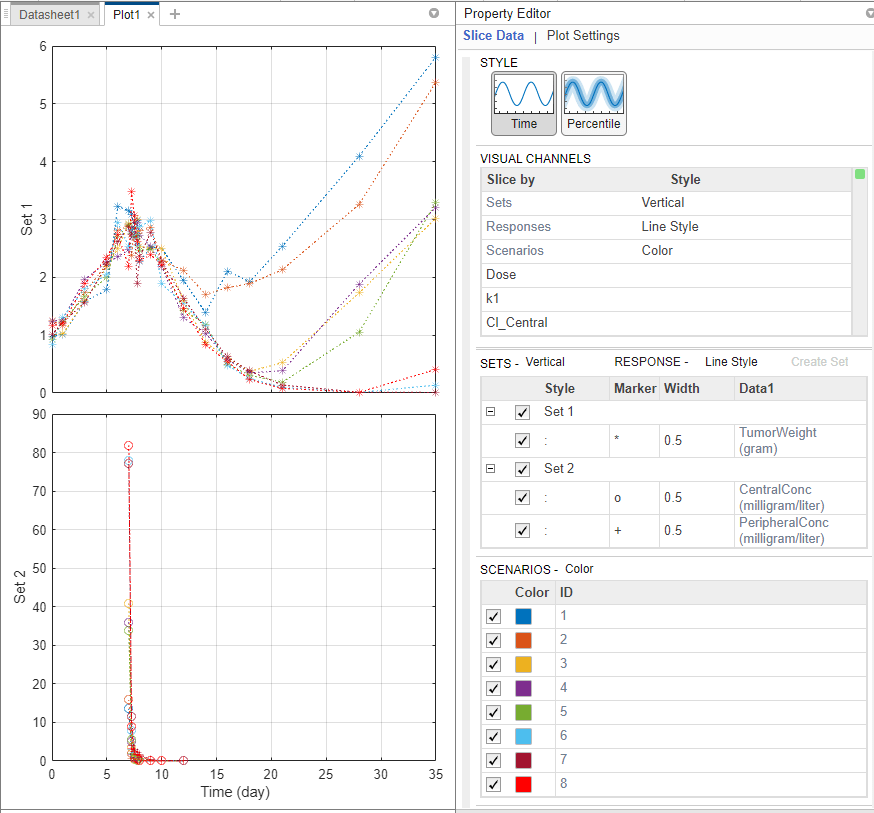
的gydF4y2Ba视觉通道gydF4y2Ba表现在包含gydF4y2Ba集gydF4y2Ba.该表是一个汇总表,包含当前在图中出现的所有切片变量及其对应的图样式。在当前的图中,切片变量是gydF4y2Ba集gydF4y2Ba,gydF4y2Ba反应gydF4y2Ba,gydF4y2Ba场景gydF4y2Ba.gydF4y2Ba
提示gydF4y2Ba
可以使用不同的切片变量对数据进行切片。每个切片变量以不同的视觉效果出现在图中gydF4y2Ba风格gydF4y2Ba(或通道),如颜色、线条样式和轴线位置。切片变量可以表示数据的属性,例如响应或场景(即组或模拟运行)。切片变量也可以是与场景或组相关的协变量或参数值。默认情况下,应用程序为绘制数据中的不同响应变量和不同场景提供切片变量。您可以为响应集和相关参数或协变量添加其他视觉样式(或通道)。gydF4y2Ba
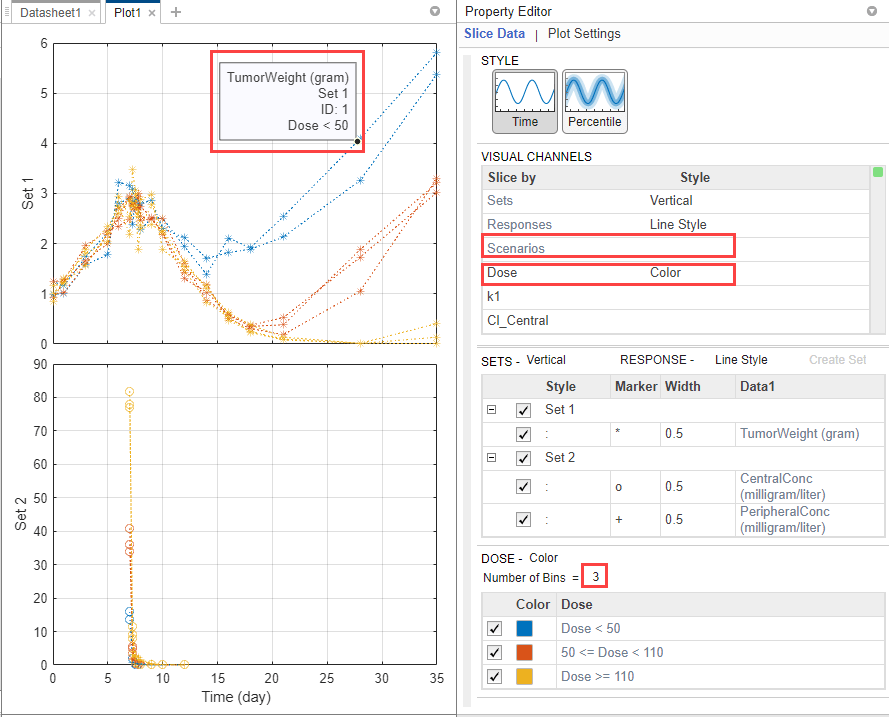
您还可以根据患者接受的不同剂量对反应进行分组。有三种不同的剂量组:30mg, 75mg和150mg。gydF4y2Ba
在gydF4y2Ba视觉通道gydF4y2Ba表,gydF4y2Ba剂量gydF4y2Ba行,双击空单元格并选择gydF4y2Ba
颜色gydF4y2Ba.出现红色指示符是因为另一个切片变量(gydF4y2Ba场景gydF4y2Ba)有着相同的情节风格。清除样式(视觉通道)gydF4y2Ba场景gydF4y2Ba通过选择空的。gydF4y2Ba在gydF4y2Ba剂量gydF4y2Ba表格,应用程序已经自动分类剂量量。集gydF4y2Ba数量的垃圾箱gydF4y2Ba来gydF4y2Ba
3.gydF4y2Ba.你现在可以看到剂量对肿瘤大小有影响。剂量越高,肿瘤越小。gydF4y2Ba您还可以通过显示每一行的数据提示来查询相应的剂量组。新闻gydF4y2BaCtrlgydF4y2Ba点击一条蓝线显示它的数据提示。删除它,gydF4y2BaCtrlgydF4y2Ba+gydF4y2Ba点击gydF4y2Ba还是在同一行的任何地方。gydF4y2Ba
执行非分区分析(NCA)gydF4y2Ba
使用药物药代动力学数据,你可以估计NCA参数。NCA是模型不可知的,可以在没有任何潜在假设的情况下洞察药物的药代动力学。在将模型校准到数据时,您可以使用一些NCA结果作为初始估计,如本示例后面所讨论的那样。有关可用NCA参数列表及其公式的详细信息,请参见gydF4y2BaNoncompartmental分析gydF4y2Ba.gydF4y2Ba
NCA程序设置gydF4y2Ba
在gydF4y2Ba首页gydF4y2Ba选项卡上,选择gydF4y2Ba程序gydF4y2Ba>gydF4y2BaNon-Compartmental分析gydF4y2Ba.一项新计划(gydF4y2BaProgram1gydF4y2Ba)出现。gydF4y2Ba
的gydF4y2Ba数据gydF4y2Ba设置gydF4y2Ba第一步定义用于NCA分析的数据集。在本例中,程序将自动进行选择gydF4y2BaData1gydF4y2Ba.gydF4y2Ba

的gydF4y2BaNCAgydF4y2Ba执行gydF4y2BaStep定义数据列关联和算法细节。在gydF4y2Ba定义gydF4y2Ba表,设置gydF4y2Ba浓度gydF4y2Ba来gydF4y2Ba
CentralConcgydF4y2Ba.保持其他设置不变。gydF4y2Ba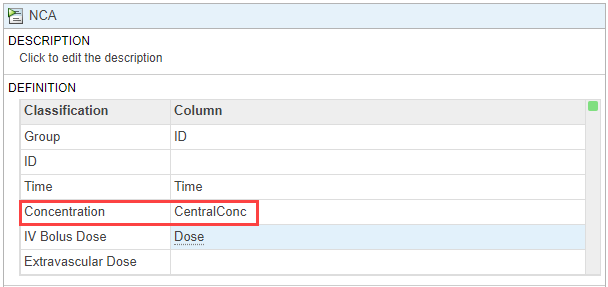
在gydF4y2Ba首页gydF4y2Ba选项卡上,单击gydF4y2Ba运行gydF4y2Ba.gydF4y2Ba
一旦NCA分析完成,应用程序将打开一个包含结果的新数据表。gydF4y2Ba

程序还将结果保存在gydF4y2BaLastRungydF4y2Ba默认为程序的文件夹。以访问结果gydF4y2Ba浏览器gydF4y2Ba窗格中,展开gydF4y2BaProgram1gydF4y2Ba文件夹中。然后展开gydF4y2BaLastRungydF4y2Ba文件夹中。NCA结果存储在名为gydF4y2Ba结果gydF4y2Ba.NCA计算参数请参见gydF4y2BaNoncompartmental分析gydF4y2Ba.gydF4y2Ba
导出结果到MATLAB工作区gydF4y2Ba
可以将NCA结果导出到MATLAB中gydF4y2Ba®gydF4y2Ba工作区,并在命令行执行进一步的数据分析。gydF4y2Ba
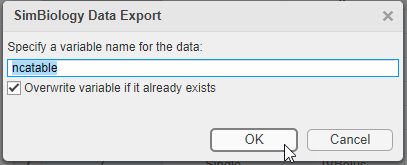
右键单击gydF4y2Ba结果gydF4y2Ba.选择gydF4y2Ba导出数据到MATLAB工作区gydF4y2Ba.gydF4y2Ba

的gydF4y2BaSimBiology数据导出gydF4y2Ba对话框打开。将变量的名称更改为gydF4y2BancatablegydF4y2Ba.点击gydF4y2Ba好吧gydF4y2Ba.gydF4y2Ba
在您将数据导出到MATLAB工作区之后,您可以在命令行中分析数据。例如,您可以从NCA数据计算平均药物清除率,并将其作为模型参数值。gydF4y2Ba
利用非线性回归估计模型参数gydF4y2Ba
SimBiology根据实验数据提供了不同的回归技术来估计模型参数。这个例子详细说明了使用非线性回归方法的步骤gydF4y2BalsqnonlingydF4y2Ba(需要优化工具箱™)使模型适合于数据。如果你没有优化工具箱,应用程序使用gydF4y2BafminsearchgydF4y2Ba代替。在本例中,只估计了PK/PD模型的部分参数,即:gydF4y2Bak1gydF4y2Ba,gydF4y2BaL0gydF4y2Ba,gydF4y2BaL1gydF4y2Ba,gydF4y2BaCl_CentralgydF4y2Ba,gydF4y2Bak12的gydF4y2Ba,gydF4y2Bak21里面gydF4y2Ba.gydF4y2Ba
符合程序的设置gydF4y2Ba
从gydF4y2Ba首页gydF4y2Ba选项卡上,选择gydF4y2Ba程序gydF4y2Ba>gydF4y2Ba合适的数据gydF4y2Ba.一项新计划(gydF4y2BaProgram2gydF4y2Ba)出现在一个新的选项卡上。的gydF4y2Ba数据gydF4y2Ba而且gydF4y2Ba模型gydF4y2Ba步骤已预先填充gydF4y2BaData1gydF4y2Ba而且gydF4y2Ba肿瘤的生长模型gydF4y2Ba,分别。gydF4y2Ba
默认情况下,gydF4y2Ba适合gydF4y2BaStep在拟合完成后自动生成图。控件顶部的情节图标可以禁用情节生成gydF4y2Ba适合gydF4y2Ba现在是程序步骤。这些情节将在后面的示例中讨论。gydF4y2Ba

在gydF4y2Ba数据地图gydF4y2Ba表中,定义模型组件和来自输入数据的数据列之间的映射。gydF4y2Ba
的gydF4y2Ba集团gydF4y2Barow标识数据中的哪一列是分组变量,例如患者id。gydF4y2Ba
的gydF4y2Ba独立的gydF4y2Ba行标识数据中的哪一列是自变量,如时间。gydF4y2Ba
的gydF4y2Ba响应gydF4y2Ba行标识哪个响应或度量数据列对应于哪个模型组件。如果有多个响应数据,可以通过单击来添加更多的响应行gydF4y2Ba响应gydF4y2Ba按钮的底部gydF4y2Ba数据地图gydF4y2Ba表格要从表中删除响应,右键单击并选择gydF4y2Ba删除gydF4y2Ba.gydF4y2Ba
的gydF4y2Ba量的数据gydF4y2Ba行定义数据中的哪列映射到作为剂量目标的哪个模型组件。如果有多个剂量列,则可以通过单击gydF4y2Ba剂量gydF4y2Ba按钮。gydF4y2Ba
的gydF4y2Ba变量的数据gydF4y2Ba行定义数据中的哪一列包含模型组件的可选参数值。单击gydF4y2Ba变体gydF4y2Ba按钮以查看该行或添加更多变体。gydF4y2Ba
请注意gydF4y2Ba
在本例中,应用程序使用输入数据的数据表中的分类信息并映射gydF4y2BaIDgydF4y2Ba类定义的分组变量gydF4y2Ba集团gydF4y2Ba表中的行),和gydF4y2Ba时间gydF4y2Ba列作为自变量(由gydF4y2Ba独立的gydF4y2Ba表中的行)。它还确定了gydF4y2BaCentralConcgydF4y2Ba,gydF4y2BaPeripheralConcgydF4y2Ba,gydF4y2BaTumorWeightgydF4y2Ba为响应列。gydF4y2Ba
在第一个响应行,挨着gydF4y2BaCentralConcgydF4y2Ba,双击单元格gydF4y2Ba组件gydF4y2Ba,并输入gydF4y2Ba中央。药物gydF4y2Ba作为该测量数据列的对应模型组件。gydF4y2Ba
同样,地图gydF4y2BaPeripheralConcgydF4y2Ba列gydF4y2Ba外围。药物gydF4y2Ba.gydF4y2Ba
地图gydF4y2BaTumorWeightgydF4y2Ba来gydF4y2Ba.tumor_weight肿瘤生长模型gydF4y2Ba.gydF4y2Ba
映射gydF4y2Ba剂量gydF4y2Ba列gydF4y2Ba中央。药物gydF4y2Ba以表明gydF4y2Ba药物gydF4y2Ba物种gydF4y2Ba中央gydF4y2Ba隔间正在下药。gydF4y2Ba

离开gydF4y2Ba变体和剂量设置gydF4y2Ba因为在本例中没有使用特定于组的剂量或变量。有关如何使用该表的详细信息,请参见gydF4y2Ba从数据集中使用剂量和变量模拟组gydF4y2Ba.gydF4y2Ba
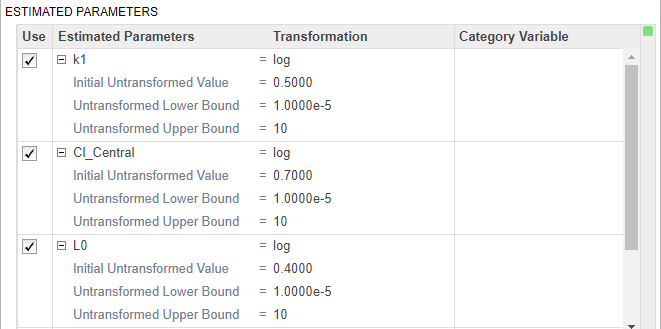
在gydF4y2Ba适合gydF4y2Ba步骤,定义待估计的模型参数gydF4y2Ba估计参数gydF4y2Ba表格控件中的空单元格gydF4y2Ba估计参数gydF4y2Ba列和类型gydF4y2Bak1gydF4y2Ba.该应用程序显示具有匹配名称的模型组件。选择gydF4y2Ba
k1gydF4y2Ba从列表中。gydF4y2Ba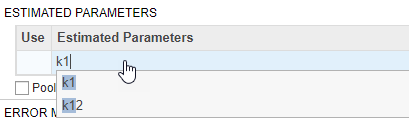
默认情况下,根据转换指示对参数进行对数转换gydF4y2Ba日志gydF4y2Ba.你可以把这个变换变成没有变换gydF4y2Ba
没有一个gydF4y2Ba,gydF4y2BaprobitgydF4y2Ba,或gydF4y2Ba分对数gydF4y2Ba转换。有关详细信息,请参见gydF4y2Ba参数转换gydF4y2Ba.对于本例,保持默认的日志转换,因为它通常可以提高收敛性。的gydF4y2Ba初始Untransformed值gydF4y2Ba自动设置为模型值0.5。gydF4y2Ba强制生物参数保持阳性gydF4y2BaUntransformed下界gydF4y2Ba而且gydF4y2BaUntransformed上界gydF4y2Ba作为gydF4y2Ba
1 e-5gydF4y2Ba而且gydF4y2Ba10gydF4y2Ba,分别。gydF4y2Ba类似地,添加以下参数:gydF4y2BaCl_CentralgydF4y2Ba,gydF4y2BaL0gydF4y2Ba,gydF4y2BaL1gydF4y2Ba,gydF4y2Bak12的gydF4y2Ba,gydF4y2Bak21里面gydF4y2Ba.gydF4y2Ba
选择gydF4y2Ba池适合gydF4y2Ba估计所有患者的一组参数(群体拟合)。如果不选择gydF4y2Ba池适合gydF4y2Ba,该应用程序估计每个患者的一组参数(个体适合度)。gydF4y2Ba
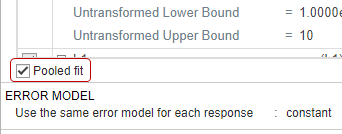
默认的误差模型是恒定误差模型。SimBiology支持常数、比例、指数和组合错误模型。有关详细信息,请参见gydF4y2Ba误差模型gydF4y2Ba.现在,使用恒定误差模型。gydF4y2Ba
保持其余配件设置不变。这些设置是gydF4y2Ba
估算方法—默认方法为gydF4y2Ba
lsqnonlingydF4y2Ba如果你有优化工具箱。如果你不这样做,应用程序使用gydF4y2BafminsearchgydF4y2Ba.gydF4y2Ba
有关更多信息,请参见gydF4y2BaSimBiology中支持的参数估计方法gydF4y2Ba.gydF4y2Ba
算法设置—估计方法的最常用选项。单击以展开该部分并查看选项。要查看每个选项的说明,请单击标题右侧的信息图标。gydF4y2Ba
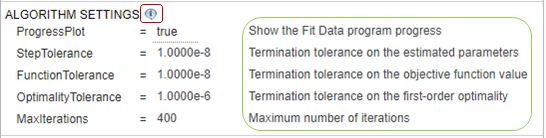
高级算法设置-评估方法的高级设置。默认情况下,该表为空。gydF4y2Ba
运行符合程序gydF4y2Ba
设置合适的选项后,可以运行gydF4y2Ba适合gydF4y2Ba的一步。gydF4y2Ba
在顶端gydF4y2Ba适合gydF4y2Ba一步,单击gydF4y2Ba运行以下程序gydF4y2Ba按钮。gydF4y2Ba
默认情况下,gydF4y2Ba适合gydF4y2BaStep在一个单独的图中显示了参数估计的进展。进度图显示了参数估计和拟合质量度量(如对数似然)的实时状态。有关详细信息,请参见gydF4y2Ba情节进展gydF4y2Ba.gydF4y2Ba
进度图显示拟合收敛。您可以关闭进度图。gydF4y2Ba
如果你正在使用gydF4y2Ba
fminsearchgydF4y2Ba,由于达到最大迭代次数,拟合可能无法收敛。你可以增加gydF4y2Ba麦克斯特gydF4y2Ba在gydF4y2Ba算法设置gydF4y2Ba但是对于本例的目的,您可以继续完成这些步骤而不这样做。gydF4y2Ba
可视化匹配的结果gydF4y2Ba
一旦参数估计完成,拟合结果将显示在一个新的数据表中。该数据表包含参数估计和与拟合质量度量相关的其他信息,如AIC和BIC,这对比较不同误差模型的性能很有用。gydF4y2Ba

除了质量统计之外,您还可以查看各种拟合图,例如实际与预测的图和剩余分布图。gydF4y2Ba
在gydF4y2Ba浏览器gydF4y2Ba窗格中,展开gydF4y2BaProgram2gydF4y2Ba>gydF4y2BaLastRungydF4y2Ba,其中包含gydF4y2Ba结果gydF4y2Ba而且gydF4y2BasimdataIgydF4y2Ba.gydF4y2Ba结果gydF4y2Ba包含估计的参数值和拟合统计信息。gydF4y2BasimdataIgydF4y2Ba包含使用估计参数值的每个个体(患者或组)的模拟模型响应。gydF4y2Ba
点击gydF4y2Ba结果gydF4y2Ba.控件中自动列出可用的拟合图gydF4y2Ba情节gydF4y2Ba上节gydF4y2Ba首页gydF4y2Ba选项卡。然后选择gydF4y2Bavs Pred行动gydF4y2Ba从列表中。gydF4y2Ba
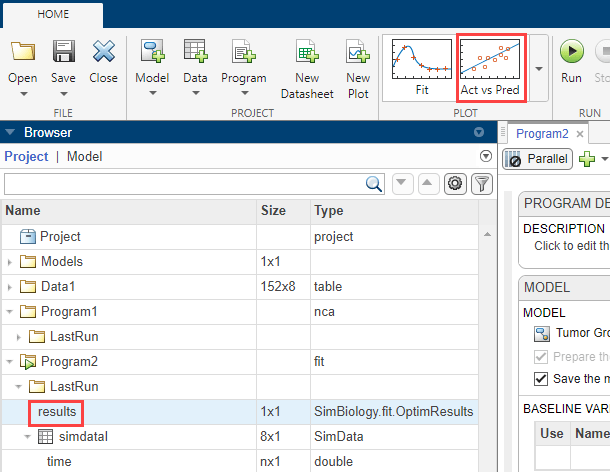
实际的和预测的图形显示在一个单独的选项卡上。预测的反应标在gydF4y2BaxgydF4y2Ba-轴和观测(实验)响应绘制在gydF4y2BaygydF4y2Ba设在。gydF4y2Ba
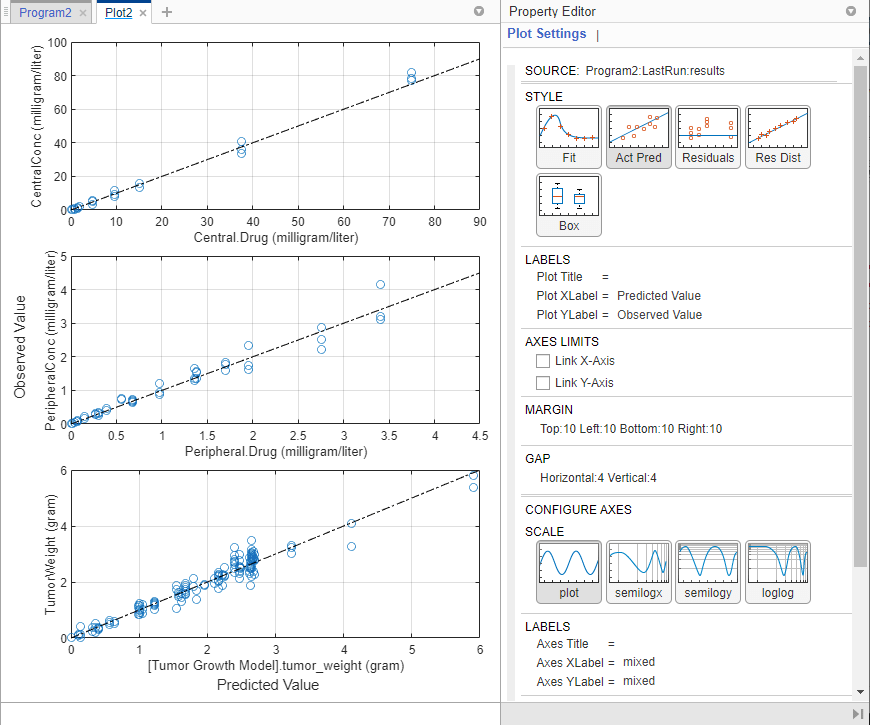
控件中的一个图形,可以将图形更改为其他支持的图形gydF4y2Ba风格gydF4y2Ba部分的gydF4y2Ba属性编辑器gydF4y2Ba.如果希望新绘图在其单独的选项卡上,并且不想重用现有绘图选项卡,请从gydF4y2Ba情节gydF4y2Ba上节gydF4y2Ba首页gydF4y2Ba选项卡。gydF4y2Ba
通过选择将地块更改为残差分布地块gydF4y2BaRes DistgydF4y2Ba在gydF4y2Ba风格gydF4y2Ba部分。gydF4y2Ba

该图显示了每个响应的残差是否呈正态分布。在残差的理想正态概率图中,残差沿横贯图的对角线排列,直方图表示正态拟合。然而,从图中,三种反应的残差,尤其gydF4y2BaCentralConcgydF4y2Ba而且gydF4y2BaPeripheralConcgydF4y2Ba,似乎不是正态分布。这可能是常数误差模型假设不正确的一个迹象。gydF4y2Ba
比较不同的误差模型gydF4y2Ba
下面的步骤展示了如何将误差模型更改为指数误差模型以再次拟合数据,并比较两种不同误差模型的拟合统计信息。gydF4y2Ba
保存结果。gydF4y2Ba在再次使用指数误差模型拟合数据之前,将恒定误差模型的结果保存在单独的文件夹中。否则,默认情况下,程序将覆盖gydF4y2BaLastRungydF4y2Ba文件夹每次运行适合。gydF4y2Ba
右键单击gydF4y2BaLastRungydF4y2Ba文件夹中的fit程序gydF4y2Ba浏览器gydF4y2Ba窗格。gydF4y2Ba
选择gydF4y2Ba
保存数据gydF4y2Ba.gydF4y2Ba在gydF4y2Ba保存数据gydF4y2Ba对话框中,输入gydF4y2Ba
fit_constantgydF4y2Ba作为数据名。gydF4y2Ba
用指数误差模型重新运行拟合。gydF4y2Ba保存数据后,可以使用不同的错误模型重新运行fit程序。gydF4y2Ba
返回到fit程序gydF4y2BaProgram2gydF4y2Ba选项卡。在gydF4y2Ba误差模型gydF4y2Ba部分中,选择gydF4y2Ba指数gydF4y2Ba.gydF4y2Ba

在顶端gydF4y2Ba适合gydF4y2Ba一步,单击gydF4y2Ba运行以下程序gydF4y2Ba按钮。gydF4y2Ba

在匹配完成后关闭进度图。gydF4y2Ba
如果您关闭前一个数据表(gydF4y2BaDatasheet3gydF4y2Ba),其中包含来自上一个适合的统计数据,重新打开数据表。要这样做,在gydF4y2Ba浏览器gydF4y2Ba窗格中,展开gydF4y2BaProgram2gydF4y2Ba>gydF4y2Bafit_constantgydF4y2Ba.然后双击gydF4y2BaDatasheet3gydF4y2Ba.gydF4y2Ba
从gydF4y2BaLastRungydF4y2Ba文件夹,拖gydF4y2Ba结果gydF4y2Ba到gydF4y2BaDatasheet3gydF4y2Ba.新列(gydF4y2BaProgram2_LastRungydF4y2Ba),其中包含最新的拟合结果会被添加到先前的拟合结果旁边(gydF4y2BaProgram2_fit_constantgydF4y2Ba).gydF4y2Ba

表格更新如下。gydF4y2Ba

的gydF4y2Ba统计数据gydF4y2Ba表比较了适合的质量措施。通过比较可知,采用指数误差模型拟合的AIC和BIC均小于先前拟合的AIC和BIC。这说明指数误差模型比常数误差模型更能拟合数据。指数误差模型的对数似然值越大,也表明其拟合度越好。gydF4y2Ba
接下来,看看残差分布图。点击gydF4y2Ba结果gydF4y2Ba从gydF4y2BaLastRungydF4y2Ba文件夹中。然后单击gydF4y2Ba剩余距离gydF4y2Ba从gydF4y2Ba情节gydF4y2Ba上节gydF4y2Ba首页gydF4y2Ba选项卡。gydF4y2Ba
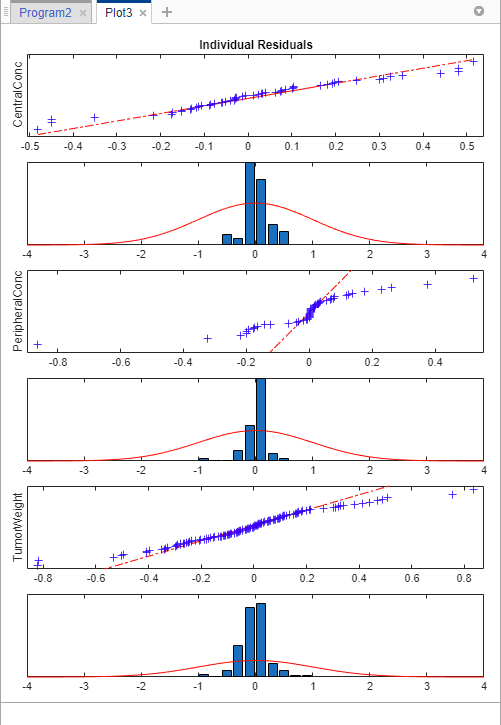
与常误差模型的残差分布相比,指数误差模型的残差分布更接近正态分布,说明指数误差模型对数据的拟合更好。gydF4y2Ba
计算置信区间gydF4y2Ba
另一种评估拟合结果质量的方法是计算估计参数和模型预测的95%置信区间——即使用估计参数的模型模拟结果。这一步需要统计和机器学习工具箱™。gydF4y2Ba
单击gydF4y2BaProgram2gydF4y2Ba选项卡。单击左上角的绿色(+)图标并选择gydF4y2Ba
置信区间gydF4y2Ba.一个gydF4y2Ba置信区间gydF4y2Ba步骤出现在gydF4y2Ba适合gydF4y2Ba的一步。gydF4y2Ba
在顶端gydF4y2Ba置信区间gydF4y2Ba步骤,通过单击图图标禁用图的自动生成。对于这两个gydF4y2Ba参数的置信区间gydF4y2Ba而且gydF4y2Ba预测的置信区间gydF4y2Ba,使用默认方法gydF4y2Ba
高斯gydF4y2Ba而且gydF4y2Ba95%gydF4y2Ba信心水平。单击gydF4y2Ba运行以下程序gydF4y2Ba按钮计算置信区间。gydF4y2Ba
对于参数置信区间,支持的方法为gydF4y2Ba高斯gydF4y2Ba,gydF4y2BaprofileLikelihoodgydF4y2Ba,gydF4y2Ba引导gydF4y2Ba.gydF4y2Ba
对于预测置信区间,支持的方法为gydF4y2Ba高斯gydF4y2Ba而且gydF4y2Ba引导gydF4y2Ba.gydF4y2Ba
一旦完成,结果被存储为gydF4y2BaparameterCIgydF4y2Ba而且gydF4y2BapredictionCIgydF4y2Ba在gydF4y2BaLastRungydF4y2Ba程序的文件夹。gydF4y2BaparameterCIgydF4y2Ba包含估计参数的95%置信区间。gydF4y2BapredictionCIgydF4y2Ba包含模型预测的95%置信区间。gydF4y2Ba
绘制估计参数的95%置信区间。点击gydF4y2BaparameterCIgydF4y2Ba在gydF4y2Ba浏览器gydF4y2Ba窗格中,选择gydF4y2Ba信心gydF4y2Ba在gydF4y2Ba情节gydF4y2Ba部分。gydF4y2Ba

每个估计参数的置信区间显示在一个新的图中。图表明成功计算了所有估计参数的置信区间。gydF4y2Ba
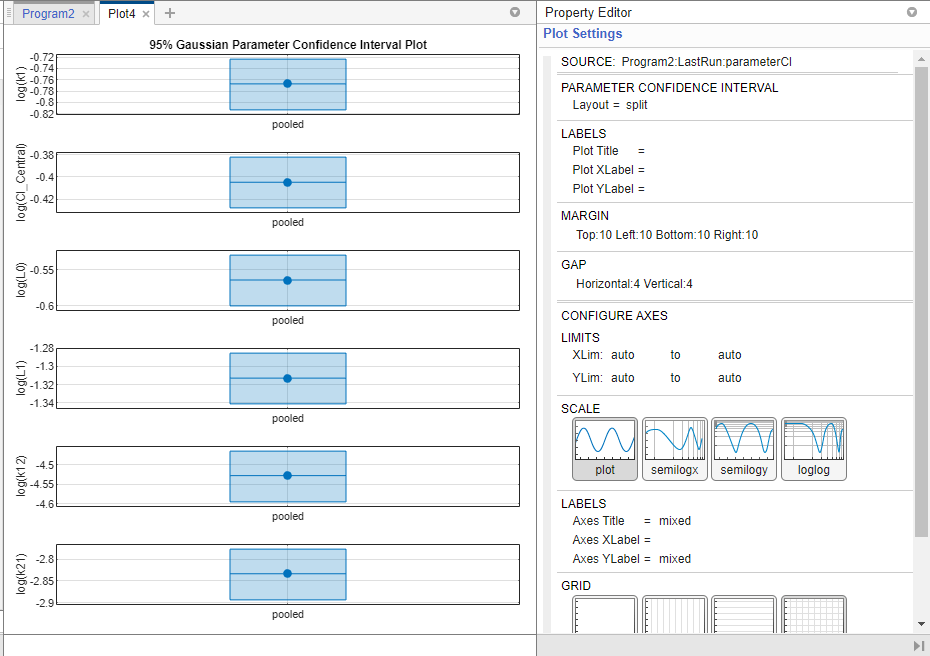
根据置信区间估计的结果(状态),应用程序绘制出不同的结果。gydF4y2Ba
如果置信区间估计的状态为gydF4y2Ba成功gydF4y2Ba(如上图所示),应用程序使用第一个默认颜色(蓝色)为每个参数估计绘制一条线和一个居中点。该应用程序还绘制了一个方框来表示置信区间。gydF4y2Ba
如果状态为gydF4y2Ba限制gydF4y2Ba或gydF4y2Ba可尊敬的gydF4y2Ba,应用程序使用第二种默认颜色(红色),并绘制一条直线、居中圆点和方框来表示置信区间。gydF4y2Ba
如果状态为gydF4y2Ba没有有价值的gydF4y2Ba在美国,该应用程序只绘制一条直线和一个以红色居中的十字。gydF4y2Ba
如果有任何经过转换的参数,其估值为0(对于gydF4y2Ba
日志gydF4y2Ba转换)和0或1(用于gydF4y2BaprobitgydF4y2Ba或gydF4y2Ba分对数gydF4y2Ba变换),没有为这些参数估计绘制置信区间。gydF4y2Ba
有关不同状态定义的详细信息,请参见gydF4y2Ba参数置信区间估计状态gydF4y2Ba.gydF4y2Ba
您也可以更改gydF4y2Ba布局gydF4y2Ba的情节gydF4y2Ba情节的设置gydF4y2Ba.gydF4y2Ba
的gydF4y2Ba
“分裂”gydF4y2Ba布局在单独的轴上显示每个参数估计的置信区间。gydF4y2Ba的gydF4y2Ba
“分组”gydF4y2Ba布局显示在一个轴上的所有置信区间,按参数估计分组。每个估计的参数都用一条垂直的黑线隔开。gydF4y2Ba
在这两种情况下,在原始拟合中定义的参数边界都用方括号标记。该应用程序使用垂直虚线来分组在共同拟合中计算的参数估计的置信区间。gydF4y2Ba
同样,绘制模型预测的95%置信区间。点击gydF4y2BapredictionCIgydF4y2Ba在gydF4y2Ba浏览器gydF4y2Ba窗格中,选择gydF4y2Ba信心gydF4y2Ba在gydF4y2Ba情节gydF4y2Ba部分。gydF4y2Ba
图中没有红色的图,表示置信区间计算成功。根据结果的不同,绘图行为也不同(gydF4y2Ba状态gydF4y2Ba)的置信区间计算。gydF4y2Ba
如果状态为gydF4y2Ba限制gydF4y2Ba或gydF4y2Ba没有有价值的gydF4y2Ba,应用程序使用第二个默认颜色(红色)绘制置信区间。gydF4y2Ba
否则,应用程序使用第一个默认颜色(蓝色)并将置信区间绘制为阴影区域(如上图所示)。gydF4y2Ba
有关详细信息,请参见gydF4y2Ba模型预测的高斯置信区间计算gydF4y2Ba而且gydF4y2BaBootstrap置信区间计算gydF4y2Ba.gydF4y2Ba
使用百分位图可视化仿真统计和叠加实验数据gydF4y2Ba
百分位图让您可视化模拟结果和统计数据,这些结果和统计数据可以与实验数据叠加。例如,您可以绘制模拟数据随时间变化的第5和第95百分位曲线,而不是查看单个时间图。您还可以可视化模拟和实验数据的平均值、标准差、最小值和最大值。有关详细信息,请参见gydF4y2Ba百分位图gydF4y2Ba.gydF4y2Ba
请注意gydF4y2Ba
如果您还没有完成前面生成所需结果的步骤,那么您可以加载已完成的项目。gydF4y2Ba
打开gydF4y2BaSimBiology模型分析gydF4y2Ba应用程序。gydF4y2Ba
点击gydF4y2Ba开放gydF4y2Ba并导航到该文件夹gydF4y2Ba
matlabrootgydF4y2Ba\ \ simbio \ \数据示例gydF4y2Ba.gydF4y2BamatlabrootgydF4y2Ba是安装MATLAB的文件夹。选择命名的项目文件gydF4y2Batumor_growth_fitPKPD_completed.sbprojgydF4y2Ba.gydF4y2Ba
在gydF4y2BaLastRungydF4y2Ba文件夹的gydF4y2BaProgram2gydF4y2Ba中,选择gydF4y2BasimDataIgydF4y2Ba>gydF4y2Ba.tumor_weight肿瘤生长模型gydF4y2Ba.gydF4y2Ba

在gydF4y2Ba首页gydF4y2Ba选项卡上,单击gydF4y2Ba百分位gydF4y2Ba.gydF4y2Ba

百分位图默认显示第5和第95百分位曲线。gydF4y2Ba

选择gydF4y2BaData1gydF4y2Ba>gydF4y2BaTumorWeightgydF4y2Ba.将其拖放到绘图中。gydF4y2Ba

默认情况下,gydF4y2Ba显示gydF4y2Ba实验数据类型自动设置为gydF4y2Ba的意思是gydF4y2Ba,表示每次测量的平均值,标准差为±1。如果希望可视化原始数据点,请双击gydF4y2Ba的意思是gydF4y2Ba并选择gydF4y2Ba原始数据gydF4y2Ba.对于本例,保留gydF4y2Ba的意思是gydF4y2Ba显示。gydF4y2Ba
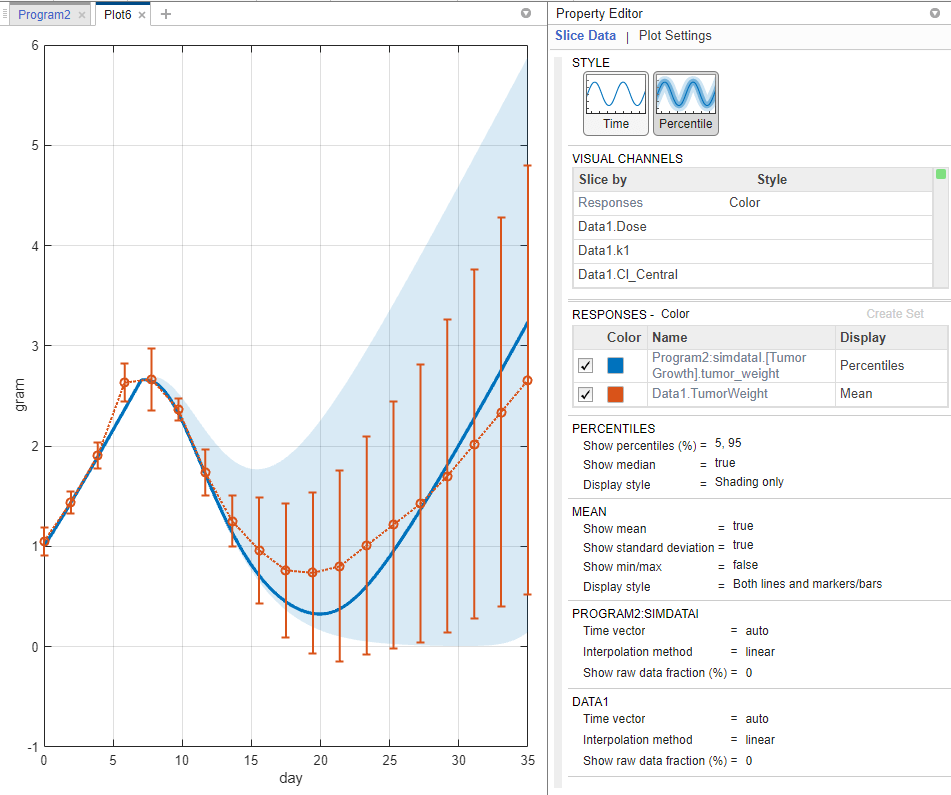
在gydF4y2Ba百分位数gydF4y2Ba的部分,gydF4y2Ba显示百分比(%)gydF4y2Ba选项,输入gydF4y2Ba
80gydF4y2Ba显示第十和八十百分位。gydF4y2Ba改变gydF4y2Ba显示风格gydF4y2Ba来gydF4y2Ba
线条和阴影gydF4y2Ba.gydF4y2Ba
在gydF4y2Ba的意思是gydF4y2Ba截面,通过设置显示每次的最小和最大响应数据点gydF4y2Ba显示最小/最大gydF4y2Ba来gydF4y2Ba
真正的gydF4y2Ba.gydF4y2Ba
在gydF4y2BaData1gydF4y2Ba部分,改变gydF4y2Ba显示原始数据百分比(%)gydF4y2Ba来gydF4y2Ba
One hundred.gydF4y2Ba以显示所有潜在的原始数据点。您也可以输入自定义百分比数字。gydF4y2Ba
百分比图更新如下。星号(gydF4y2Ba
*gydF4y2Ba)表示插值后计算得到的最小值和最大值,点(gydF4y2Ba.gydF4y2Ba)表示原始数据点。因为这些最小值和最大值是根据插值的公共时间向量计算的,所以这些值与原始数据的值并不完全匹配。有关详细信息,请参见gydF4y2Ba意思是选择gydF4y2Ba.gydF4y2Ba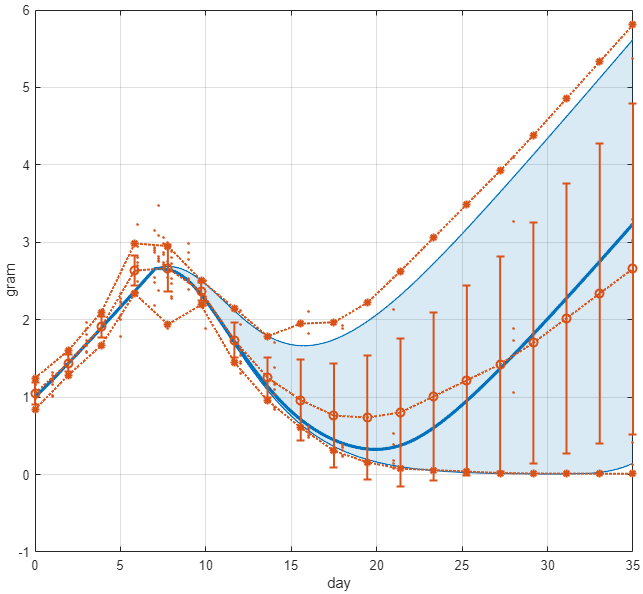
在进行参数估计之后,您可以将模型值设置为参数估计并执行其他分析。例如,您可以使用gydF4y2Ba敏感性分析gydF4y2Ba并通过改变敏感参数来研究模型的可变性gydF4y2Ba虚拟病人gydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
[1]gydF4y2Ba西米奥尼,莫尼卡,保罗·马格尼,克里斯蒂亚诺·卡米娅,朱塞佩·德·尼可拉奥,瓦尔特·克罗奇,恩里克·佩森蒂,马西米利亚诺·日尔曼尼,伊塔洛·波吉西,和毛里齐奥·罗凯蒂。给药后异种移植瘤模型中肿瘤生长动力学的预测药代动力学-药效学建模gydF4y2Ba癌症研究gydF4y2Ba64年,没有。3(2004年2月1日):1094-1101。gydF4y2Ba




































