模拟葡萄糖-胰岛素反应
这个例子展示了如何在SimBiology®中使用基于生理学的正常和糖尿病人的葡萄糖-胰岛素系统模型来模拟和分析一个模型。
本示例需要统计和机器学习工具箱™和优化工具箱™。
参考文献
葡萄糖-胰岛素系统的膳食模拟模型。C.达拉曼,R.A.里扎,C.科贝利。IEEE生物医学工程汇刊(2007) 54(10), 1740 - 1749。
口服葡萄糖吸收系统模型:金标准数据验证。C.达拉曼,M.卡米列里,C.科贝利。IEEE生物医学工程汇刊(2006) 53(12), 2472 - 2478。
目标
实现一个SimBiology的葡萄糖-胰岛素反应模型。
模拟正常和受损(糖尿病)受试者一餐或多餐后的葡萄糖-胰岛素反应。
执行参数估计
sbiofit用强迫函数策略。
背景
在他们2007年的出版物中,Dalla Man等人开发了一个人类餐后葡萄糖-胰岛素反应的模型。该模型用常微分方程描述系统的动力学。作者使用他们的模型模拟了一顿或多顿饭后的葡萄糖-胰岛素反应,包括正常人和各种胰岛素受损的人。损伤用参数值和初始条件的交替集表示。
我们实现了SimBiology模型,m1由:
将Dalla Man等人(2007)的模型方程转化为反应、规则和事件。
将模型组织成两个单元,一个单元用于葡萄糖相关的物种和反应(命名为
葡萄糖的外表)和一个关于胰岛素相关的物种和反应(命名为胰岛素分泌).利用模型方程及表1和图1中的参数值和初始条件。
包括Dalla Man等人(2006)提出的胃排空率方程。
根据Dalla Man等人(2007)的规定,设置所有物种、隔间和参数的单位,这允许使用单位转换来模拟SimBiology模型。(注意SimBiology还支持使用无量纲参数,通过设置它们的
ValueUnits财产无量纲的)。设置配置集
TimeUnits来小时,因为模拟时间超过7或24小时。以1公斤体重为基础,对原模型中按体重归一化的物种和参数进行变换。这样做使得物种的数量或浓度成为单位,正如《SimBiology》所要求的那样。
我们将SimBiology模型中的胰岛素损伤表示为具有以下名称的变量对象:
2型糖尿病
低胰岛素敏感性
β细胞反应性高
β细胞反应性低
高胰岛素敏感性
我们将SimBiology模型中的食物表示为剂量对象:
一个剂量名叫
一顿简单的饭菜代表模拟开始时一餐78克葡萄糖。一个剂量名叫
日常生活代表一天的膳食价值,相对于从午夜开始的模拟:早餐是45克葡萄糖在模拟时间的8小时(早上8点),午餐是70克葡萄糖在12小时(中午),晚餐是70克葡萄糖在20小时(晚上8点)。
SimBiology模型示意图如下:

设置
加载模型。
sbioloadproject (“insulindemo”,“m1”)
抑制模拟过程中发出的信息警告。
warnSettings =警告(“关闭”,“SimBiology: DimAnalysisNotDone_MatlabFcn_Dimensionless”);
模拟正常人的葡萄糖-胰岛素反应
选择一顿简单的饭菜剂量对象并显示其属性。
mealDose = sbioselect (m1,“名字”,“一餐”);get (mealDose)
ans =结构体字段:Amount: 78 Interval: 0 Rate: 0 RepeatCount: 0 StartTime: 0 Active: 0 AmountUnits: 'gram' DurationParameterName: " EventMode: 'stop' LagParameterName: " RateUnits: " TargetName: 'Dose' TimeUnits: 'hour' Name: 'Single Meal' Parent: [1x1 SimBiology.]注释:"标签:"类型:'repeatdose' UserData: []
模拟7小时。
configset = getconfigset (m1,“活跃”);configset。StopTime=7;
显示模拟时间单位(和StopTime单位)。
configset。TimeUnits
ans =“小时”
模拟一个正常的受试者吃一顿饭。
normalMealSim = sbiosimulation (m1, configset, [], mealDose);
模拟2型糖尿病患者的血糖-胰岛素反应
选择2型糖尿病变体并显示其属性。
diabeticVar = sbioselect (m1,“名字”,“2型糖尿病”)
diabeticVar = SimBiology变体- 2型糖尿病(非活动)ContentIndex:类型:名称:属性:值:1参数血浆容量…1.49 2 parameter k1 0.042 3 parameter k2 0.071 4 parameter血浆体积…0.04 5参数m1 0.379 6参数m2 0.673 7参数m4 0.269 8参数m5 0.0526 9参数m6 0.8118 10参数肝外…0.6 11参数kmax 0.0465 12参数kmin 0.0076 13参数kbs 0.023 14参数kgri 0.0465 15参数f 0.9 16参数a 6e-05 17参数b 0.68 18参数c 0.00023 19参数d 0.09 20参数kp1 3.09 21参数kp2 0.0007 22参数kp3 0.005 23参数kp4 0.0786 24参数ki 0.0066 25参数[Ins Ind Glu U…1 26参数Vm0值4.65 27参数Vmx值0.034 28参数Vmx值466.21 29参数p2U值0.084 30参数K值0.99 31参数alpha值0.013 32参数beta值0.05 33参数gamma值0.5 34参数ke1值0.0007 35参数ke2值269 36参数基等离子体G…值164.18 37 parameter basic Plasma I…价值54.81
模拟2型糖尿病患者的一顿饭。
diabticmealsim = sbiosimulation (m1, configset, diabeticVar, mealDose);
比较模拟的最重要输出的结果。
血浆葡萄糖(物种
等离子体Glu浓缩的)血浆胰岛素(物种
血浆Ins浓缩的)内源性葡萄糖生成(参数
Glu刺激)葡萄糖出现率(参数
Glu出现率)葡萄糖利用率(参数
Glu跑龙套)胰岛素分泌(参数
Ins Secr)
outputNames = {“等离子Glu浓缩”,“血浆Ins浓缩的”,“Glu刺激”,...“Glu出现率”,“Glu Util”,“Ins Secr”};图;为i = 1: numl (outputNames) subplot(2,3, i);[tNormal, yNormal] = normalMealSim.selectbyname(outputNames{i});[tdiabetes, ydiabetes] = diabticmealsim .selectbyname(outputNames{i});plot(tNormal, yNormal,“- - -”,...糖尿病患者,糖尿病患者,“——”);%注释数据outputParam = sbioselect (m1,“名字”我,outputNames {});标题(outputNames{我});包含(的时间(小时));如果比较字符串(outputParam。类型,“参数”) ylabel (outputParam.ValueUnits);其他的ylabel (outputParam.InitialAmountUnits);结束xlim ([0 7]);%添加传奇如果I == 3 legend({“正常”,“糖尿病”},“位置”,“最佳”);结束结束

注意血浆中葡萄糖和胰岛素的高浓度,以及葡萄糖利用和胰岛素分泌的持续时间延长。
模拟普通受试者一日三餐
设置模拟StopTime24小时。
configset。StopTime=24;
选择每日膳食剂量。
dayDose = sbioselect (m1,“名字”,“日常生活”);
模拟一个正常受试者的三餐。
normalDaySim = sbiosimulation (m1, configset, [], dayDose);
模拟残疾受试者一日三餐
模拟以下损伤组合:
损伤1:胰岛素敏感性低
损伤2:β细胞反应性高的损伤1
损伤3:β细胞反应性低
损伤4:损伤3伴高胰岛素敏感性
将缺陷存储在单元格数组中。
impairVars {1} = sbioselect (m1,“名字”,“低胰岛素敏感性”) ;(impairVars impairVars {2} = {1},...sbioselect (m1,“名字”,“β细胞的高反应性”));impairVars {3} = sbioselect (m1,“名字”,“β细胞反应性低”) ;(impairVars impairVars {4} = {3},...sbioselect (m1,“名字”,“高胰岛素敏感性”));
模拟每个障碍。
为i = 1:4 dyssims (i) = sbiosimulate(m1, configset, dysvars {i}, dayDose);结束
比较血糖和血浆胰岛素结果。
图;outputNames = {“等离子Glu浓缩”,“血浆Ins浓缩的”};legendLabels = {{“正常”},...{“ins =β\ ',“ins +β\”},...{“β= Ins - \”,“+ Ins -β\”}};yLimits = [80 240;0 500];为i = 1:numel(outputNames) [tNormal, yNormal] = selectbyname(normalDaySim, outputNames{i});[tImpair, y损害]= selectbyname(受损sims, outputNames{i});%绘制正常Subplot (2,3,3 *i-2);情节(tNormal yNormal,“b -”);xlim (24 [0]);ylim (yLimits(我,:));包含(的时间(小时));传奇(legendLabels {1},“位置”,“西北”);图示低胰岛素Subplot (2,3,3 *i-1);情节(tImpair {1}, yImpair {1},“g——”, tImpair {2}, yImpair {2},“:”);xlim (24 [0]);ylim (yLimits(我,:));包含(的时间(小时));传奇(legendLabels {2},“位置”,“西北”);标题(outputNames{我});% Plot Low BetaSubplot (2,3,3 *i);情节(tImpair {3}, yImpair {3},c -。, tImpair {4}, yImpair {4},“m -”);xlim (24 [0]);ylim (yLimits(我,:));包含(的时间(小时));传奇(legendLabels {3},“位置”,“西北”);结束

注意低胰岛素敏感性(虚线绿色) )或β -细胞敏感性低(虚线青色线, )导致血糖浓度增加和延长(图中最上面一排)。一个系统的低灵敏度可以由另一个系统的高灵敏度部分补偿。例如,低胰岛素敏感性和高β细胞敏感性(虚线红色, )的结果是相对正常的血糖浓度(图的第一排)。然而,在这种情况下,产生的血浆胰岛素浓度非常高(图的最下面一排)。
参数估计方法
本文采用强制函数策略对模型的不同子系统进行参数估计,而不是同时对整个模型进行参数估计。这种方法需要额外的实验数据作为子模型的“输入”。在拟合过程中,输入数据决定了输入物种的动态。(在完整模型中,输入的动力学是由微分方程确定的。)在SimBiology术语中,您可以将强制函数实现为重复分配规则,该规则控制作为模型子系统输入的物种或参数的值。在接下来的章节中,我们使用SimBiology的参数拟合功能来完善作者报告的参数值。
葡萄糖表象胃肠模型的拟合nlinfit
胃肠道模型表示食物中的葡萄糖是如何通过胃、肠、肠运输,然后被血浆吸收的。这个子系统的输入是一顿饭中的葡萄糖量,输出是血浆中葡萄糖的出现率。然而,由于作者报告的数值与参数和模拟结果不一致,我们也估计了餐粉尺寸。因为这个输入只发生在模拟开始时,所以不需要强制函数。
这个函数sbiofit支持使用MATLAB™、统计和机器学习工具箱、优化工具箱和全局优化工具箱中的几种不同算法对SimBiology模型中的参数进行估计。首先,利用统计和机器学习工具箱功能估计参数nlinfit.
加载实验数据fitData = groupedData (readtable (“GlucoseData.csv”,“分隔符”,”、“));设置数据上的单位fitData.Properties.VariableUnits = {...“小时”,...%的时间单位毫克/分钟的,...% GluRate单位毫克/分升的,...% PlasmaGluConc单位毫克/分钟的,...% GluUtil单位};识别哪些模型组件对应观察到的数据变量。。gastroFitObs =“[Glu出现率]= GluRate”;%估计以下参数的值:gastroFitEst = estimatedInfo ({“kmax”,“kmin”,“出租车”,“剂量”});在进行估计期间,确保参数估计总是正的。%,使用对所有参数的日志转换。[gastroFitEst。变换]=交易(“日志”);将“剂量”的初始估计值设置为报告的餐量。的%剩余的初始估计值将从%的模型。gastroFitEst(4)。我nitialValue = mealDose.Amount;生成初始参数估计的模拟数据。configset。StopTime=7; gastroInitSim = sbiosimulate(m1, mealDose);%使用|nlinfit|来拟合数据,在每次迭代中显示输出fitOptions = statset (“显示”,“通路”);[gastroFitResults, gastroFitSims] = sbiofit(m1, fitData,...gastroFitObs gastroFitEst, [],“nlinfit”, fitOptions);
迭代SSE梯度步长模数----------------------------------------------------------- 0 43.798 1 2.23537 22.9971 0.596828 2 1.65217 1.33015 0.107431 3 1.6491 0.0778864 0.0239157 4 1.64909 0.00058626 0.00224484 5 1.64909 0.0141494 0.000316232 6 1.64909 0.0141494 0.000316232 6 2.23868e-05 7 1.64909 0.0839478 0.000117587 8 1.64909 0.0472672 9.25494e-07迭代终止:SSE的相对变化小于OPTIONS。TolFun
拟合数据使用fminunc
现在,使用优化工具箱函数估计参数fminunc.
拟合数据,绘制每次迭代的目标函数。fitOptions2 = optimoptions (“fminunc”,“PlotFcns”, @optimplotfval);[gastroFitResults(2), gastroFitSims(2)] = sbiofit(m1, fitData,...gastroFitObs gastroFitEst, [],“fminunc”, fitOptions2);

比较拟合前后的仿真结果。
gastroSims = selectbyname([gastroInitSim gastroFitSims],“Glu出现率”);图;情节(gastroSims(1)。时间,gastroSims(1)。数据,“- - -”,...gastroSims(2)。时间,gastroSims(2)。数据,“——”,...gastroSims(3)。时间,gastroSims(3)。数据,“-”。,...fitData。时间,fitData。GluRate,“x”);包含(的时间(小时));ylabel (毫克/分钟的);传奇(“报告”,“估计(nlinfit)”,...“估计(fminunc)”,“实验”);标题(葡萄糖的外表适合的);

绘制参数值相对于报告值的变化。
图;.ParameterEstimates.Estimate fitResults = [gastroFitResults (1)...gastroFitResults (2) .ParameterEstimates.Estimate];kmax、kmin和kkabs的初始值来自模型。gastroFitInitValues = [get(sbioselect(m1,“名字”,“kmax”),“价值”)得到(sbioselect (m1,“名字”,“kmin”),“价值”)得到(sbioselect (m1,“名字”,“出租车”),“价值”) gastroFitEst(4)。InitialValue];relFitChange = fitResults ./ [gastroFitInitValues gastroFitInitValues] - 1;酒吧(relFitChange);甘氨胆酸ax =;斧子。XTickLabel = {gastroFitEst.Name};ylabel (“估值相对变动”);标题(“比较报告和估计的胃肠参数值”);传奇({“nlinfit”,“fminunc”},“位置”,“北”)

注意,如果餐粉尺寸(剂量)显著大于报告的参数kmax比报道的要大得多,而且出租车比报道的要小。
肌肉和脂肪组织葡萄糖利用模型的拟合
肌肉和脂肪组织模型表示葡萄糖在体内是如何被利用的。这个子系统的“输入”是血浆中胰岛素的浓度(血浆Ins浓缩的),内源性葡萄糖产生(Glu刺激)和葡萄糖的出现率(Glu出现率).“输出”是血浆中葡萄糖的浓度(等离子体Glu浓缩的)和葡萄糖利用率(Glu跑龙套).
因为输入是时间的函数,它们需要作为强制函数来实现。的值血浆Ins浓缩的,Glu刺激,Glu出现率由重复分配控制,调用函数对报告的实验值进行线性插值。当使用这些函数控制一个类或参数时,必须使用于设置其值的任何其他规则处于不活动状态。为了便于选择这些规则,规则Name属性包含有意义的名称。
为“输入”创建强制函数:%血浆胰岛素PlasmaInsRule = sbioselect (m1,“名字”,“等离子体合成定义”);PlasmaInsForcingFcn = sbioselect (m1,“名字”,“等离子体in Conc强迫函数”)
plasmains强迫fcn = SimBiology规则数组索引:RuleType: Rule: 1 repeatedAssignment [Plasma Ins Conc] =[皮摩尔每升]*PlasmaInsulin(时间/[一小时])
PlasmaInsRule。积极= false;PlasmaInsForcingFcn。积极= true;内源性葡萄糖生成(Glu Prod)GluProdRule = sbioselect (m1,“名字”,“Glu刺激定义”);GluProdForcingFcn = sbioselect (m1,“名字”,“Glu Prod强迫功能”)
gluprodforceingfcn = SimBiology规则组索引:RuleType: Rule: 1 repeatedAssignment [Glu Prod] =[毫克每分钟]*内源性葡萄糖生产(时间/[一小时])
GluProdRule。积极= false;GluProdForcingFcn。积极= true;葡萄糖出现率(葡萄糖出现率)GluRateRule = sbioselect (m1,“名字”,“Glu出现率定义”);GluRateForcingFcn = sbioselect (m1,“名字”,“Glu出现速率强制函数”)
glurateforceingfcn = SimBiology规则数组索引:RuleType: Rule: 1 repeatedAssignment [Glu出现率]=[毫克每分钟]* glucoseappearance ancerate (time/[一小时])
GluRateRule。积极= false;GluRateForcingFcn。积极= true;使用初始参数值进行模拟muscleInitSim = sbiosimulate (m1);识别哪些模型组件对应观察到的数据变量。。muscleFitObs = {'[Plasma GluConc] = PlasmaGluConc',...'[Glu Util] = GluUtil'};%估计以下参数的值:muscleFitEst = estimatedInfo ({“(等离子体体积(Glu))”,“k1”,“k2”,...“Vm0”,“Vmx”,“公里”,“p2U”});在进行估计期间,确保参数估计总是正的。%,使用对所有参数的日志转换。[muscleFitEst。变换]=交易(“日志”);%拟合数据,在每次迭代时显示输出[muscleFitResults, muscleFitSim] = sbiofit(m1, fitData,...muscleFitObs muscleFitEst, [],“nlinfit”, fitOptions);
迭代SSE梯度步骤的Norm of Norm ----------------------------------------------------------- 0 2181.52 1 1085.41 39015.9 0.610025 2 323.327 7382.05 0.430616 3 292.039 2304.89 0.0415646 4 282.959 2695.63 0.0967634 6 262.927 568.782 0.0226267.77 0.0208718 8 261.519 2277.77 0.0101182 9 258.937 1615.12 0.0149182 876.168 0.0559867 11 252.87 540.854 0.00679402 12 252.273 808.908 0.0125569 13 252.271 1159.19 0.000761151 14 252.256 518.3496.4832e-05 15 252.177 722.555 0.001689 16 252.072 231.853 0.00552735 857.883 0.0191843 18 250.906 195.796 0.00951454 19 250.774 378.47 0.0110708 20 250.773 554.932 0.0007185 21 250.767 520.599 7.67298e-05 22 250.766 306.758 2.07328e-06 23 250.766 511.073 4.55003e-07 24 250.766 498.737 1.60482e-16迭代终止:当前步骤的相对norm小于OPTIONS。TolX
绘制参数值相对于报告值的变化。
图;muscleFitInitValues = [get(sbioselect(m1,“名字”,“等离子体体积(Glu)”),“价值”)得到(sbioselect (m1,“名字”,“k1”),“价值”)得到(sbioselect (m1,“名字”,“k2”),“价值”)得到(sbioselect (m1,“名字”,“Vm0”),“价值”)得到(sbioselect (m1,“名字”,“Vmx”),“价值”)得到(sbioselect (m1,“名字”,“公里”),“价值”)得到(sbioselect (m1,“名字”,“p2U”),“价值”));栏(muscleFitResults.ParameterEstimates。Estimate ./ muscleFitInitValues - 1);甘氨胆酸ax =;斧子。XTickLabel = {muscleFitEst.Name};ylabel (“估值相对变动”);标题(“比较报告和估计的葡萄糖参数值”);

清理对模型的更改。
PlasmaInsRule。积极= true;GluProdRule。积极= true;GluRateRule。积极= true;PlasmaInsForcingFcn。积极= false;GluProdForcingFcn。积极= false;GluRateForcingFcn。积极= false;
比较拟合前后的仿真结果
muscleSims = selectbyname([muscleInitSim muscleFitSim],...{“等离子Glu浓缩”,“Glu Util”});图;情节(muscleSims(1)。时间,muscleSims (1) . data (: 1),“- - -”,...muscleSims(2)。时间,muscleSims (2) . data (: 1),“——”,...fitData。时间,fitData。PlasmaGluConc,“x”);包含(的时间(小时));ylabel (“mg / dl”);传奇(“初始(模拟)”,“估计(模拟)”,“实验”);标题(“血浆葡萄糖配合”);

图;情节(muscleSims(1)。时间,muscleSims (1) . data (:, 2),“- - -”,...muscleSims(2)。时间,muscleSims (2) . data (:, 2),“——”,...fitData。时间,fitData。GluUtil,“x”);包含(的时间(小时));ylabel (毫克/分钟的);传奇(“初始(模拟)”,“估计(模拟)”,“实验”);标题(葡萄糖利用合适的);
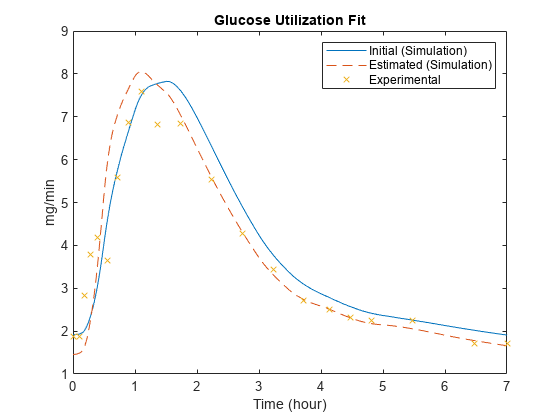
注意显著增加一些参数,例如Vmx,可以更好地拟合后期血糖浓度。
清理
恢复设置的警告。
警告(warnSettings);
结论
SimBiology包含几个特性,有助于实现和模拟葡萄糖-胰岛素系统的复杂模型。反应、事件和规则提供了描述系统动态的自然方法。单位转换允许以方便的单位指定物种和参数,并确保模型的尺寸一致性。剂量对象是描述模型重复输入的一种简单方法,例如本例中的每日用餐计划。SimBiology还为模拟和参数估计等分析任务提供内置支持。
